ARN polymérase
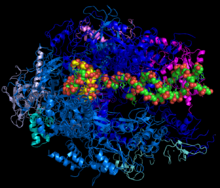
Structure de l'ARN polymérase II eucaryote, complexée à un ADN matrice (vert), avec un brin d'ARN en cours de synthèse (jaune)
| N° EC | EC |
|---|---|
| N° CAS |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | Structures |
| GO | AmiGO / EGO |
L'ARN polymérase est un complexe enzymatique responsable de la synthèse de l'acide ribonucléique, ou ARN, à partir d'une matrice d'ADN. Ce processus biologique, présent dans toutes les cellules, s'appelle la transcription. Chez les eucaryotes, il existe essentiellement trois ARN polymérases — l'ARN polymérase I, l'ARN polymérase II et l'ARN polymérase III tandis que chez les procaryotes il n'existe qu'une seule ARN polymérase.
Sommaire
1 Rôle de l'ARN polymérase
1.1 Le promoteur
1.2 Chez les archées
1.3 Chez les bactéries
1.4 Chez les eucaryotes
1.4.1 ARN polymérases I
1.4.2 ARN polymérases II
1.4.3 ARN polymérases III
1.4.4 ARN polymérases IV
1.5 Chez les virus
2 Notes et références
3 Voir aussi
3.1 Articles connexes
Rôle de l'ARN polymérase |
Elle assure, ou plutôt catalyse la synthèse des quatre types d'ARN : l'ARN ribosomique, l'ARN messager, l'ARN de transfert, et les petits ARN : micro-ARN, petits ARN interférents, ARNsn du spliceosome (ARNsn U1, ARNsn U2, ARNsn U4, ARNsn U5, ARNsn U6), ARN de voûte, etc.
L'ARN polymérase transcrit un premier brin d'ARN que l'on nomme « transcrit primaire » à partir d'un des deux brin du duplexe d'ADN utilisé comme matrice. Pour cela elle se fixe sur une séquence particulière appelée promoteur de transcription.
Le promoteur |
Il existe des différences dans les séquences des promoteurs, à la fois selon les gènes, en fonction de l'ARN polymérase concernée, mais aussi d'une espèce à l'autre. Pour la synthèse des ARN messagers, le promoteur comporte en général deux régions ou "boites" conservées : une boite située juste en amont du site de démarrage de la transcription, appelée boîte de Pribnow chez les bactéries et boîte TATA chez les eucaryotes. Elle est centrée à 10 nucléotides en amont du gène chez les procaryotes, et à 25 nucléotides en amont chez les eucaryotes. La boîte TATA peut parfois être remplacée par la boîte GC (GGGCGG) située 50 à 60 nucléotides en amont du gène. Il existe également une deuxième région plus en amont : la boîte CAAT située 70 à 80 nucléotides en amont du gène, chez les eucaryotes et la boite "-35" chez les bactéries. Cette seconde boite contribue de réguler l'intensité de transcription du gène et donc la quantité d'ARN synthétisé.
Lorsqu'elle se fixe sur la boîte TATA (ou GC), l'ARN polymérase II s'associe avec différentes protéines appelées facteurs de transcription (abrégés TF en anglais, pour transcription factor) pour former une particule d'initiation. Il existe des facteurs de transcription généraux et des facteurs de transcription spécifiques de certains gènes.
Les ARN polymérases I et III eucaryotes reconnaissent des promoteurs de nature différente de ceux de l'ARN polymérase II.
La liaison entre l'ADN et l'ARN polymérase au niveau du promoteur permet d'une part d'ouvrir la double hélice sur une quinzaine de paires de bases et donc de rendre disponible un des deux brins d'ADN pour permettre son utilisation comme matrice. Ceci permet de catalyser l'insertion des quelques premiers ribonucléotides triphosphates pour amorcer la synthèse du brin d'ARN. Ensuite l'ARN polymérase commence sa progression sur l'ADN matrice, en quittant le promoteur. La polymérisation de l'ARN devient alors très processive.
Chez les archées |
Les archées utilisent une seule ARN polymérase pour transcrire leurs gènes, tout comme les bactéries, même si leur enzyme montre une forte conservation structurale avec l'ARN polymérase II des eucaryotes[1].
L'histoire de la découverte de l’ARN polymérase des archées est assez récente. La première analyse chez les archées a été faite en 1971, quand l'ARN polymérase de Halobacterium cutirubrum, une halophile extrême, a été isolée et purifiée[2].
Les récentes structures radiocristallographiques de l’ARN polymérase de Sulfolobus solfataricus et de Sulfolobus shibatae révèlent l'image complète de cette enzyme et ont permis l’identification de 13 sous-unités dans l’enzyme archéenne[1],[3].
Les ARN polymérases dans les trois domaines du vivant partagent une architecture et des mécanismes moléculaires communs, ce qui suggère une relation évolutive ancestrale[4]. En outre, les différences entre l’ARN polymérase archéenne et celle des autres procaryotes sont en accord avec les travaux de phylogénie moléculaire de Carl Woese.
Chez les bactéries |
Chez les bactéries, il n'existe qu'une seule ARN polymérase, polymère d'un poids moléculaire d'environ 500 kDa, découverte dans les années 1960 chez la bactérie Escherichia coli, sa composition est la suivante :
- Le cœur de l’enzyme ARN polymérase = α2ββ’ω
- Holo-enzyme = α2ββ’ωσ
Les deux sous-unités α permettent de maintenir le brin non-transcrit à l'écart. Constituent la base sur laquelle s'organise le reste de la molécule. Les sous-unités β’ et β contiennent le site actif et permettent de lier le substrat de l'enzyme. La sous-unité σ se détache au début de la transcription : elle permet de reconnaître le promoteur. La sous-unité ω participe à la liaison de l'enzyme sur la matrice d'ADN : elle permet de maintenir la sous-unité β’ dans sa bonne conformation et de la recruter pour former une enzyme fonctionnelle. La sous-unité σ permet de diminuer d'un facteur 10 000 l'affinité de l'ARN polymérase pour des séquences quelconques d'ADN, et au contraire augmente l'affinité de cette même enzyme pour le site promoteur.
L'ARN polymérase associée au facteur σ (sigma) , qui est une sous-unité de l'enzyme, est capable d'initier la transcription car le facteur sigma se fixe à l'ADN en reconnaissant de manière spécifique une séquence de ce dernier, le promoteur qui est situé en amont du site d'initiation de la transcription. Cela permet à l'ARN polymérase de se fixer sur le premier nucléotide à transcrire.
Il est à noter que le facteur σ détecte deux séquences : une région à -35 nucléotides et une autre à -10 , on appelle ces deux régions des "boites".
Une fois que les premiers ribonucléotides ont été synthétisés, le facteur sigma devenu inutile se détache. À partir de ce moment la transcription s'opère à un rythme de 40 nucléotides par seconde environ.
La fin de la transcription est signalée par des terminateurs de transcription. Il en existe deux types :
- Une structure en tige-boucle sur l'ARN messager, suivie d'une série de résidus d'uridine. C'est le cas le plus fréquent. Elle est appelée terminaison intrinsèque.
- Le facteur ρ (Rho) qui est une enzyme à forte activité hélicase ATP-dépendante. C'est une terminaison Rho-dépendante.
Chez les eucaryotes |
ARN polymérases I |
Les ARN polymérases I permettent la synthèse des ARN ribosomiques (tous sauf l'ARNr 5S).
Déroulement de la transcription :
Elle possède 2 régions promotrices : PC (de -40 à +20) et UCE (Upstream Central Element) (de -107 à -180). Le facteur UBF1 se fixe sur ces régions. Puis c'est SL1 qui se fixe sur UBF1, permettant à l'ARN Polymérase I de se fixer à son tour. SL1 joue le rôle de sigma chez les procaryotes. L'élongation peut alors débuter[5].
La transcription se fait sous forme de « sapin de noël ». Les ADN correspondant aux ARNr font partie des séquences moyennement répétées en tandem. Le système de transcription fait que la production de transcrit est très efficace. Toutes les 100 pbs, il y a une ARN polymérase qui vient se fixer. Aux extrémités des « branches » il y a fixation de protéines qui vont permettre la maturation de l’ARN[6].
ARN polymérases II |
Cette ARN polymérase possède 11 sous unités, et conserve le noyau central qui est présent dans l'ARN polymérase procaryote, comme les trois ARN polymérases eucaryotes.
L'ARN polymérase II catalyse la formation de l'ARN messager ou, plus souvent, d'un ARN prémessager. Elle catalyse également la biosynthèse des snoARN (small nucleosomal RNA), snARN, microARN (miARN) et siARN. Cette enzyme est située dans le nucléoplasme. Elle est composée de 12 sous-unités[7].
ARN polymérases III |
Les ARN polymérases III sont responsables de la synthèse d'ARN courts comme l'ARN de transfert, quelques snARN (U6) et l'ARN ribosomique 5S.
ARN polymérases IV |
Les ARN polymérases IV sont spécifiques des plantes. Elles sont spécialisées dans la transcription des petits ARN interférents.
Chez les virus |
L'ARN polymérase d'origine virale la plus connue et la plus étudiée est celle du bactériophage T7 qui catalyse la transcription de 5' en 3'. C'est un complexe enzymatique de poids moléculaire 99 kDa se liant à un promoteur très spécifique au niveau de l'ADN (TAATACGACTCACTATAG, la transcription commençant au niveau du G en 3'). L'ARN polymérase T7 est largement utilisée en biologie moléculaire pour transcrire de l'ADN cloné sur des vecteurs en partie car elle transcrit avec un taux d'erreur très faible. Elle n'est pas sensible à la rifampicine comme l'ARN polymérase bactérienne.
Les orthopoxvirus synthétisent de l'ARN via une ARN polymérase virale très proche structurellement et fonctionnellement de celles retrouvées chez les bactéries, les archées et les eucaryotes (I-V). La plupart des autres virus possèdent des polymérases non reliées à celles des autres domaines du vivant.
Il existe des virus utilisant des ARN polymérases dépendantes de l'ADN comme matrice et d'autres dépendantes de l'ARN (retrouvées chez certains virus à ARN simple ou doubles brins).
Notes et références |
Korkhin, Y., U. Unligil, O. Littlefield, P. Nelson, D. Stuart, P. Sigler, S. Bell and N. Abrescia (2009). "Evolution of Complex RNA Polymerases: The Complete Archaeal RNA Polymerase Structure." PLoS biology 7(5): e102.
Louis, B. G. and P. S. Fitt (1971). "Nucleic acid enzymology of extremely halophilic bacteria. Halobacterium cutirubrum deoxyribonucleic acid-dependent ribonucleic acid polymerase." The Biochemical journal 121(4): 621-627.
Hirata, A., B. Klein and K. Murakami (2008). "The X-ray crystal structure of RNA polymerase from Archaea." Nature 451(7180): 851-854.
Werner, F. (2007). "Structure and function of archaeal RNA polymerases." Molecular microbiology 65(6): 1395-1404.
« second year biochemistry », sur biocadmin.otago.ac.nz (consulté le 14 avril 2016)
« Figure 1 : Dysregulation of the basal RNA polymerase transcription apparatus in cancer : Nature Reviews Cancer », sur www.nature.com (consulté le 14 avril 2016)
« Structure of RNA Polymerase II », Science (consulté le 30 octobre 2009) : « RNA polymerase II (Pol II) is a large (550 kDa) complex of 12 subunits that is at the heart of the transcription mechanism. »
Voir aussi |
Outre l'ARN polymérase ADN-dépendante (assurant la transcription d'une matrice d'ADN en ARN), il existe des ARN polymérases ARN-dépendantes, fabriquant de l'ARN à partir d'une matrice d'ARN. Ces enzymes participent à la réplication de certains virus à ARN, dont le génome est composé d'ARN ; on appelle ces enzymes des réplicases.
Articles connexes |
- ADN polymérase
Acide ribonucléique (ARN)
- Portail de la biochimie
- Portail de la biologie cellulaire et moléculaire
